Протеомика
Протео́мика, междисциплинарная наука, направленная на изучение всей совокупности белков в живых организмах, их модификаций и структурно-функциональных особенностей; набор методов и технологий высокопроизводительного определения и анализа белков. Протеомика является продолжением исследования геномов (геномики), ориентированным на изучение белков как основного продукта реализации наследственной информации. Современная протеомика подразделяется на такие субдисциплины, как интерактомика, структурная протеомика, клиническая протеомика и др.
Этимология термина
Термины «протеом» и «протеомика» были предложены М. Р. Уилкинсом в 1994 г. (Wilkins. 1996). Термин «протеом» является объединением слов «белок» (protein, от греч. πρώτειος – основной), предложенного Й. Я. Берцелиусом в 1838 г. (Miquel. 1838), и «геном» (genome), предложенного Г. К. А. Винклером в 1920 г. (Winkler. 1920). В свою очередь термин «геном» (genome) происходит от ген (gene, от греч. γένος) и суффикса -ом (объединение, совокупность). Впервые суффикс -ик (-ics) в составе слова «геномика» (genomics) предложил Т. Родерик в 1986 г. В настоящее время на этимологической основе терминов «геномика» и «протеомика» и возник суффикс «омик» (-omics), который означает объединяющую дисциплину, в связи с чем возникает общий термин «омики» («омиксы»). К омикам относятся метаболомика (идентификация и количественный анализ метаболома), транскриптомика (количественный анализ экспрессии генов) и др. (Yadav. 2007).
История развития
В 1950 г. научная группа П. В. Эдмана предложила метод секвенирования пептидных последовательностей (Edman. 1950). В 1951–1955 гг. группа Ф. Сенгера расшифровала аминокислотную последовательность инсулина, чем доказала упорядоченную структуру последовательности белков (Sanger. 1949). В 1959 г. Р. Сассмен-Ялоу и С. А. Берсон разработали метод радиоиммунного анализа (Berson. 1959), который дал начало всем современным методам иммунного анализа, в том числе иммуноферментного анализа. С помощью радиоиммунного анализа можно идентифицировать конкретные белки в растворах.
Параллельно в 1961 г. М. У. Ниренберг, Й. Маттеи и другие исследователи установили правила кодирования аминокислот в молекулах рибонуклеиновой кислоты (РНК) (Nirenberg. 1961). Методы генетики позволили к 2001 г. получить рабочий черновик генома человека.
В 1970 г. У. Лэммли предложил один из основных методов разделения и идентификации белков – гель-электрофорез в денатурирующих условиях (sodium dodecyl sulfate polyacrylamide, SDS-PAGE) (Laemmli. 1970). В 1975 г. независимо друг от друга П. Г. О’Фарелл (O’Farrell. 1975) и Й. Клозе (Klose. 1975) создали основы метода двумерного электрофореза (2D-электрофореза). За счет модификаций, предложенных А. Гёрг, появилась возможность создания т. н. протеомных карт тканей, органов или целых организмов, которые стали основой анализа экспрессии генов, кодирующих соответствующие белки (Görg. 1980).
В 1981 г. М. Барбер разработал метод бомбардировки быстрыми атомами (Fast atom bombardment ... 1981), в 1984 г. Дж. Б. Фенн и М. Ямасита изобрели метод электрораспыления ионизацией (electrospray ionization, ESI), а в 1985–1988 гг. М. Карас, Ф. Хиленкамп (Matrix-assisted ultraviolet ... 1987) и Т. Коити (Protein and polymer analyses up to m/z 100 00 ... 1988) предложили метод матрично-активированной лазерной десорбции/ионизации (Matrix Assisted Laser Desorption/Ionization, MALDI), или мягкой ионизации молекул лазером. Эти методы позволяют получать ионы без разрушения молекул и дают возможность применения метода масс-спектрометрии (МС).
В 1993–1996 гг. с помощью МС идентифицированы пептиды, полученные в результате действия протеолитических ферментов (трипсина) (Pappin. 1993) (Yates. 1993). В 1994–2000 гг. разработаны первые программы, способные автоматически идентифицировать пептиды из спектров (Eng. 1994). В 1999–2001 гг. появляется скорострельная протеомика, которая объединяет методы высокоэффективной жидкостной хроматографии (ВЭЖХ) и тандемной МС (МС, в процессе которой первичные ионы разделяются на более мелкие и анализируются ещё раз, также часто называется МС/МС или МС2). Этот подход позволяет идентифицировать большее количество белков в образце.
В конце 1990-х г. А. А. Макаров изобрёл ионную ловушку Orbitrap, способную определять ионы с высокой точностью (Makarov. 2000).
В 2008 г. методами биоинформатики был сформирован первый предсказанный черновик (незавершённая сборка) протеома человека (The Universal Protein Resource (UniProt) 2009. 2009 ).
В 2010 гг. начался самый известный проект «Протеом человека» (The Human Proteome Project, HPP) (Omenn. 2012). В проекте участвуют группы исследователей из 18 стран, инициаторами проекта при этом являются 6 стран: Республика Корея, США, РФ, Швеция, Канада и Иран.
Картированию белков человека посвящены и другие проекты, например Human Proteome Map и ProteomicsDB. К маю 2022 г. в рамках этих трёх проектов найдено и описано почти 18,4 тыс. белков («Протеом человека»), 30 тыс. (Human Proteome Map) и 16,4 тыс. (ProteomicsDB).
В целом к 2022 г. идентифицировано более 93,2 % существующих белков (не учитывая изоформы и протеоформы). В базе данных Protein Data Bank (PDB) представлено 56 795 структур белков, из них уникальными являются только 3 тыс. Ежегодная конференция CASP проводит конкурс новых проектов по предсказанию пространственной структуры белков на основе сравнения результатов конкурсантов и лабораторных методов. Победителем 2020 г. стала программа AlphaFold, разработанная Google DeepMind на базе искусственного интеллекта (Callaway. 2022). Качество работы этого проекта сопоставимо с точностью лабораторных методов.
Методы протеомики
Качественные и количественные методы: ионообменная, эксклюзионная и аффинная хроматография; иммуноферментный анализ; вестерн-блот; использование аналитических, функциональных и обратно-фазовых белковых микрочипов; электрофорез белков в полиакриламидном геле (sodium dodecyl sulfate-polyacrylamide gel electrophoresis, SDS-PAGE) и 2D-электрофорез; масс-спектрометрия, в том числе методы скорострельной протеомики; метод секвенирования Эдмана; изотопная маркировка для масс-спектрометрии (isotope-coded affinity tag, iCAT); маркировка аминокислот стабильными изотопами (stable isotope labeling by amino acids in cell culture, SILAC); изобарическая маркировка (isobaric tags for relative and absolute quantitation, iTRAQ).
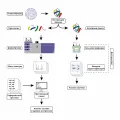 Классические методы протеомного анализа: ВЭЖХ/МС/МС и вестерн-блот. ВЭЖХ – высокоэффективная жидкостная хроматография, МС – масс-спектрометрия.
Классические методы протеомного анализа: ВЭЖХ/МС/МС и вестерн-блот. ВЭЖХ – высокоэффективная жидкостная хроматография, МС – масс-спектрометрия.Структурные методы: рентгеновский структурный анализ (РСА); ядерный магнитный резонанс (ЯМР); криоэлектронная микроскопия (Крио-ЭМ).
Биоинформационные методы: структурная биоинформатика; протеомный поиск; анализ баз данных.
Генетические методы: yeast two hybrid (Y2H); рекомбинантные технологии; нокауты.
Методы протеомики предназначены для получения информации о первичной (т. е. аминокислотной последовательности) и третичной (пространственной) структуре белка; уровне его экспрессии в разных тканях и при различных условиях; функциях; белок-белковых взаимодействиях; типичных или патологических аминокислотных заменах; модификациях.
Определение функций белка основано на генетических экспериментах с нокаутированием, гибридизацией и модификацией генов исследуемых белков в модельных организмах и культурах клеток. В частности, метод yeast two-hybrid screening (Y2H) позволяет идентифицировать белок-белковое взаимодействие за счёт активации экспрессии генов дрожжей. Активация регулируется исследуемыми генетически модифицированными белками. Y2H является одним из двух основных методов интерактомики (вместе с МС).
Белковый состав, или протеомный профиль, организма, ткани или отдельной клетки можно составить, используя скорострельную протеомику, 2D-электрофорез или белковые микрочипы. Белковые микрочипы являются самыми быстрыми методами идентификации белковых профилей и, в частности, могут быть использованы для диагностики патологий.
Уровень экспрессии белков определяется с помощью метода масс-спектрометрии с предварительным разделением белковых смесей из тканей с использованием 2D-электрофореза или ВЭЖХ. Идентификация белков осуществляется путём нахождения протеотипических пептидов (пептиды, однозначно характеризующие исследуемый белок, обычно включают в себя от 9 до 13 аминокислот, для идентификации белка требуется минимум 2 пептида). Обычно достаточно использования классической МС для идентификации белков и их уровней экспрессии. Однако для более точного определения уровней экспрессии при поиске новых маркеров различных патологий или мишеней для лекарств используют высокоточные методы iCAT, iTRAQ и SILAC.
Протеомный поиск – основной метод идентификации пептидов в результатах МС. С помощью методов биоинформатики создаётся база для поиска, которая обычно включает в себя последовательности всех известных белков или их протеолитических пептидов, а также пептидов, содержащих замену (если необходимо), и заведомо неправильных последовательностей аминокислот, образованных обратным прочтением реальных последовательностей (target-decoy). На основе поисковой базы предсказываются виртуальные спектры пептидов. Далее спектр, полученный из эксперимента, накладывается на виртуальный, и происходит идентификация совпадений. Для тех спектров, которые совпали с поисковой базой, указываются вероятный пептид и уровень его представленности (уровень экспрессии).
Третичная (пространственная) структура белков в настоящее время определяется с помощью методов РСА, ЯМР и крио-ЭМ, а также предсказывается с использованием методов структурной биоинформатики. Из пространственной структуры получают важную информацию о свойствах и функциях белков, а также о возможности их использования в качестве мишеней лекарств.
Для предсказания пространственной структуры белка используют методы моделирования: методы ab initio, построение по гомологии и др. С помощью методов ab initio пытаются предсказать процесс упаковки белка на основе термодинамических закономерностей, а также результаты фолдинга. Метод построения по гомологии является главенствующим в протеомике, однако он ограничен количеством знаний о структурах белков. Проект AlphaFold и сервис SwissModel являются одними из способов моделирования белков по гомологии, однако они не лишены недостатков, и для большинства белков, структура гомологов которых неизвестна, предсказание оказывается непригодным к использованию. Методы молекулярной динамики, квантовой механики позволяют моделировать некоторое подобие пространственной упаковки, однако чаще всего результаты далеки от реальности.
Несмотря на неполную информацию о протеомах организмов, на данный момент методы протеомики активно используются для оценки размеров протеомов различных организмов; определения функций идентифицированных белков; получения протеомных профилей и идентификации отдельных пептидов в норме и при патологии; изучения влияния различных соединений на белковый состав и количество белков в клетке; составления функциональных генетических, белковых, метаболических и других биологических сетей; диагностики заболеваний; идентификации и классификации видов и штаммов организмов; разработки новых фармацевтических препаратов; анализа пищевой продукции.
Научные журналы
Ведущими научными журналами по теме протеомики являются Molecular and Cellular Proteomics, Journal of Proteome Research, Journal of Proteomics, Proteomics, Expert Reviews Proteomics.
Базы биологических данных
Постоянно пополняются основные базы данных протеомных исследований: YRC PDR (данные экспериментов на клетках дрожжей с аннотацией белковых функций и взаимодействий); PRIDE (архивная база данных спектров, полученных методом масс-спектрометрии); PeptideAtlas (база аннотированных белков в экспериментальных данных, в том числе с указанием тканеспецифичной экспрессии); UniProt (база, собирающая всю информацию о белках из курируемой базы Swiss-Prot с более чем 500 тыс. белков и автоматически аннотированной базы TrEMBL с более чем 230 млн белков); PDB (база данных структур белков и других биополимеров).
